Fusion 分析
前言
IMPORTANT
Fusion 模块专注于单细胞转录组融合基因检测分析,通过结合 STAR-Fusion 算法和单细胞转录组数据,实现细胞水平的融合基因鉴定、可视化和功能分析。流程支持 human 和 mouse 物种,能够从原始 fastq 文件或预处理数据中识别融合事件,并在单细胞分辨率上进行展示。
基因融合作为重要的分子事件,在肿瘤发生、细胞分化和疾病进展中扮演着关键角色。SeekSoul Online 云平台的 Fusion 模块提供了从原始数据处理到结果可视化的完整解决方案,能够准确识别融合基因、评估其在不同细胞群体中的分布,并提供详细的生物学注释信息。
Fusion 分析理论基础
核心原理
- 数据预处理:对输入的 fastq 文件进行质控和过滤,去除低质量 reads 和接头序列,确保后续分析的准确性。
- 融合基因检测:使用 STAR-Fusion 算法进行融合事件识别,该算法结合了 STAR 比对器的高灵敏度和专用的融合过滤流程,能够有效识别各种类型的基因融合。
- 融合验证与注释:对检测到的融合事件进行验证,评估其可靠性,并提供丰富的注释信息,包括断点位置、支持 reads 数、融合类型等。
- 单细胞整合分析:将融合基因检测结果与单细胞转录组数据整合,识别携带特定融合的细胞群体。
- 可视化与报告生成:生成 UMAP/TSNE 可视化图,直观展示融合基因在不同细胞群体中的分布,并生成包含详细分析结果的 HTML 报告。
技术特点
| 特点 | 说明 | 优势 |
|---|---|---|
| STAR-Fusion 算法 | 基于 STAR 比对器,能够高效识别融合转录本。 | 高灵敏度、低假阳性率。 |
| 单细胞水平解析 | 将融合事件映射到特定细胞群体。 | 提供细胞异质性视角。 |
| 多物种支持 | 支持 human 和 mouse 基因组分析。 | 适用范围广。 |
| 丰富的注释信息 | 提供断点位置、融合类型、功能注释等。 | 便于生物学解读。 |
| 直观的可视化 | UMAP/TSNE 散点图展示融合基因分布。 | 便于结果解释和展示。 |
关键统计指标
- JunctionReadCount:包含在融合连接位点处,一条 read 可以拆分匹配到两侧融合基因的 reads 数量。
- SpanningFragCount:包含融合连接的 reads 数量,该 reads 的 R1 端和 R2 端对应基因不同。
- FFPM:支持融合的 reads 的标准化结果,即每百万总 reads 数的融合量。
- LargeAnchorSupport:在假定断点两侧是否存在 reads 的较长碱基序列 (≥25) 匹配,缺少 LargeAnchorSupport 的融合基因通常为假阳性。
- PROT_FUSION_TYPE:蛋白质融合类型,如 INFRAME (框内融合) 等。
SeekSoul Online 云平台操作指南
分析前准备
CAUTION
- 输入数据应为成对的 fastq 文件(R1 和 R2),确保文件命名规范,便于系统正确识别样本信息。
- 确保选择正确的物种(human 或 mouse),这将影响参考基因组的选择和后续分析的准确性。
- 对于大规模数据,建议先进行数据质量评估,确保数据质量满足分析要求。
参数详解
| 界面参数 | 说明 | 备注 |
|---|---|---|
| 任务名称 | 英文开头,可含中文/数字/下划线 | 用于报告抬头与任务跟踪 |
| 样本信息 | 输入样本名称、R1 和 R2 fastq 文件路径 | 支持多个样本并行分析 |
| 物种 | human / mouse | 决定使用的参考基因组 |
| 备注 | 自定义文本 | 记录分析背景信息 |

结果解读
结果目录速览
| 路径 | 内容 | 说明 |
|---|---|---|
output/results/fusion/ | 融合基因检测原始结果。 | 包含 STAR-Fusion 输出文件。 |
output/results/plots/ | 融合基因可视化图表。 | UMAP/TSNE 散点图。 |
output/results/meta/ | 元数据信息。 | 细胞注释和统计数据。 |
report/ | HTML 报告目录。 | 包含完整分析结果的报告。 |
融合基因表格解读
| 列名 | 说明 | 重要性 |
|---|---|---|
#FusionName | 融合基因名称 | 主键标识符 |
JunctionReadCount | 连接位点支持 reads 数 | 评估可信度 |
SpanningFragCount | 跨融合片段 reads 数 | 评估可信度 |
LeftGene/RightGene | 融合涉及的两个基因 | 功能分析基础 |
LeftBreakpoint/RightBreakpoint | 融合断点位置 | 结构分析关键 |
LargeAnchorSupport | 长锚点支持情况 | 过滤假阳性重要指标 |
FFPM | 融合 reads 标准化值 | 定量表达水平 |
PROT_FUSION_TYPE | 蛋白质融合类型 | 功能预测依据 |
关键图表示例
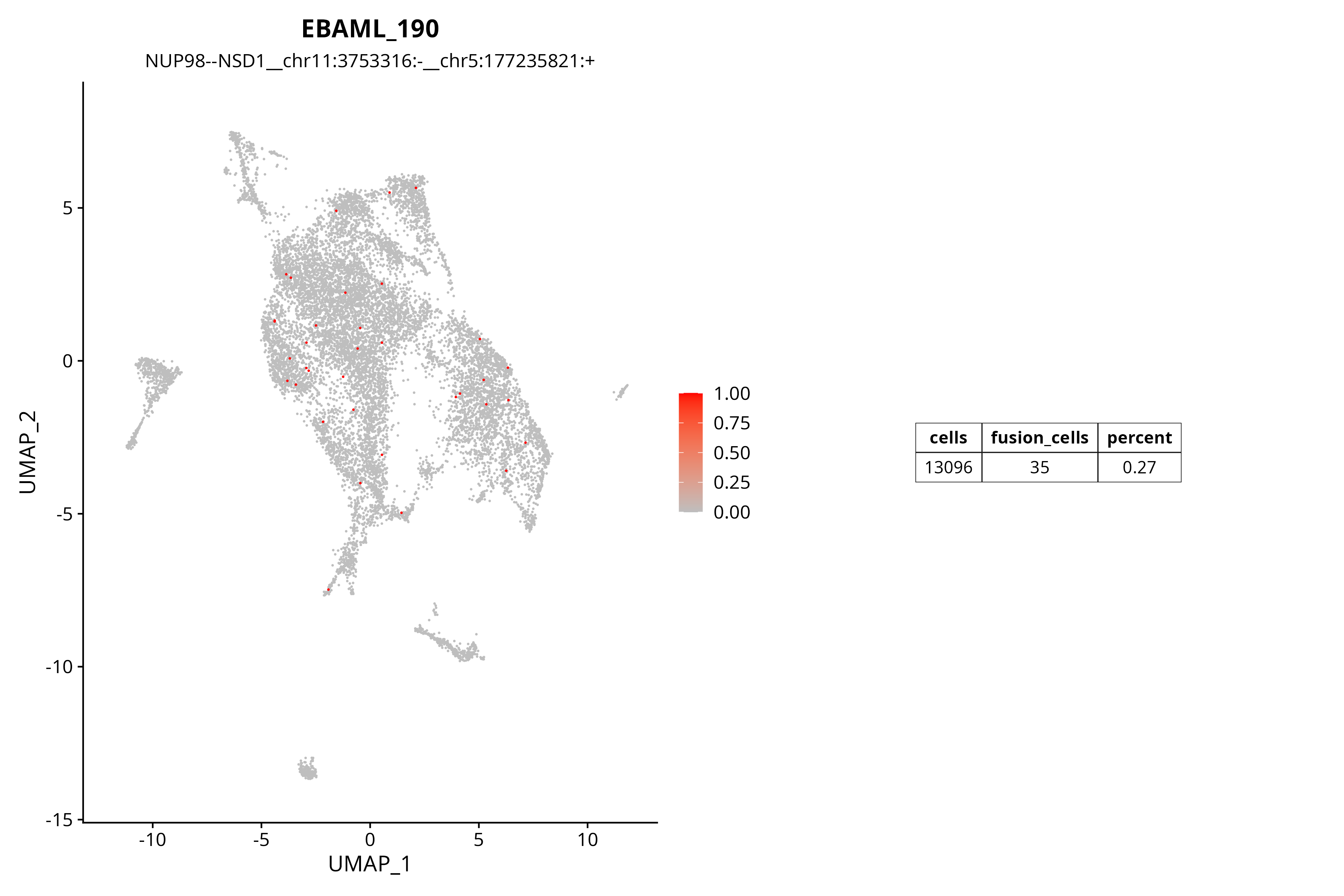
融合基因 UMAP 分布图
下图展示了 NUP98--NSD1 融合基因在单细胞群体中的分布情况。颜色深浅表示 UMI 计数,红色表示高表达该融合的细胞。
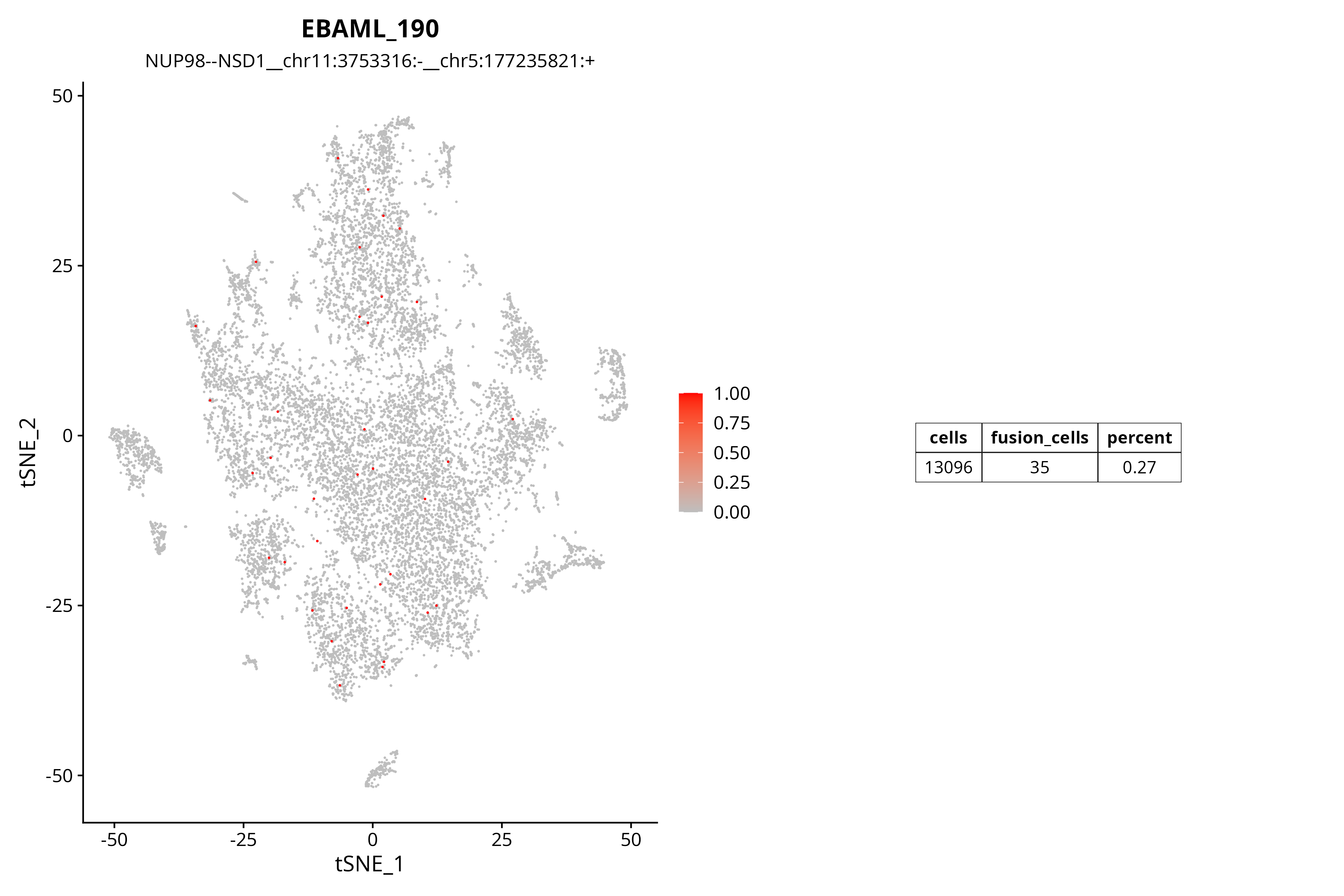
融合基因 TSNE 分布图
TSNE 图提供了另一种可视化视角,有助于识别融合基因在不同细胞亚群中的分布模式。
生物学意义解读
- 功能分类:根据融合基因的注释信息(如 NUP98:Oncogene),评估其在细胞生物学过程中的潜在作用。
- 细胞特异性:分析融合基因在不同细胞类型中的分布模式,确定其表达的细胞特异性。
- 融合类型分析:根据融合断点位置和蛋白质融合类型,预测融合蛋白的结构和功能特性。
- 数据库关联:通过与 Mitelman、ChimerKB 等数据库的比对,获取已知融合事件的临床和生物学信息。
案例参考
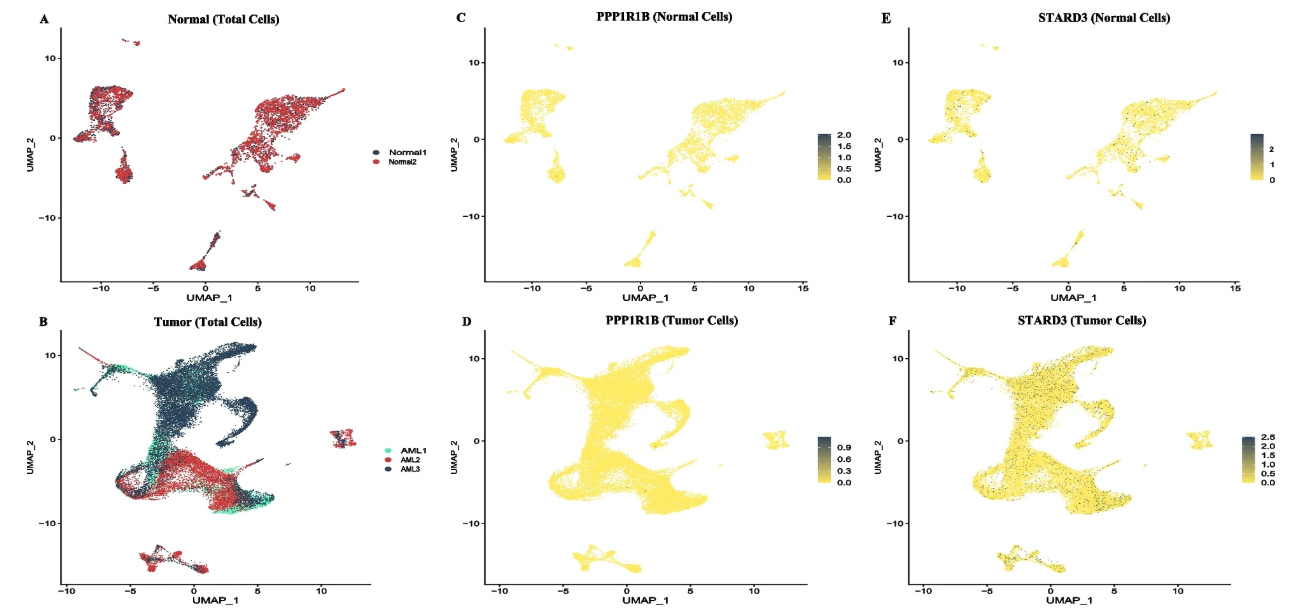
Fusion 模块的分析结果可广泛应用于多种研究场景,特别是在白血病研究领域具有重要价值。以 PPP1R1B::STARD3 融合为例:
- 新型融合基因发现:PPP1R1B::STARD3 融合是首次在急性髓系白血病 (AML) 中报道的融合事件,通过单细胞 Fusion 分析,可以精确定位携带该融合的细胞群体,了解其在肿瘤异质性中的分布模式。
- 治疗靶点发现:该融合基因涉及胆固醇代谢和 PI3K/AKT 信号通路,STARD3 参与细胞内胆固醇运输,PPP1R1B 在多种实体瘤中作为癌基因发挥作用,其融合产物可能成为新的治疗靶点。
- 疾病诊断标志物:该融合基因在 AML 患者中特异性表达,而在 B 细胞急性淋巴细胞白血病 (B-ALL) 中未见报道,具有作为 AML 诊断标志物的潜力。
推荐的实践路径是:
- 融合筛选:利用融合基因表格中的支持 reads 数、FFPM 等指标,筛选高可信度的融合事件。
- 细胞定位:通过 UMAP/TSNE 可视化,确定融合基因在不同细胞群体中的分布情况。
- 功能注释:结合数据库注释信息,评估融合基因的生物学意义和潜在功能影响。
- 验证实验:对重要的融合事件进行后续实验验证,如 RT-PCR、FISH 等技术。
注意事项与最佳实践
WARNING
融合基因检测结果中可能存在假阳性,需要结合多个指标进行筛选,如 JunctionReadCount、LargeAnchorSupport 等。对于重要的融合事件,建议进行实验验证。
- 数据质量控制:确保输入数据质量良好,通过 fastp 等工具进行质控,过滤低质量 reads,提高融合检测的准确性。
- 参数优化:根据具体研究需求,合理调整分析参数,如物种选择、过滤阈值等。
- 结果验证:对检测到的重要融合事件,建议使用 RT-PCR、Sanger 测序等方法进行实验验证。
- 生物学解读:结合现有文献和数据库信息,对融合事件进行深入的生物学解读,避免过度解读或误读。
常见问题 (FAQ)
Q:如何评估融合基因的可信度? A:主要参考以下指标:高 JunctionReadCount 和 SpanningFragCount、存在 LargeAnchorSupport、正常的 Left/RightBreakEntropy 值(通常在 1.5-2.0 之间)、具有已知的生物学功能注释等。
Q:为什么有些融合事件在 UMAP/TSNE 图上分布较散? A:这可能是由于融合事件在多个细胞群体中都有表达,或者样本中存在细胞异质性较高的情况。需要结合具体的生物学背景进行解释。
Q:如何区分真正的融合基因和假阳性? A:除了关注统计指标外,还可以参考以下几点:融合基因是否在多个公共数据库中有记录、断点是否位于内含子区域且符合 GT-AG 剪接信号、融合产物是否具有潜在的生物学功能等。
Q:分析结果中发现了大量线粒体基因相关的融合,这是正常的吗? A:线粒体基因 (MT- 开头) 参与的融合事件较为常见,但很多可能是技术原因导致的假阳性。建议结合注释信息和生物学知识进行筛选,重点关注与已知疾病相关的融合事件。
参考资料
[1] HAAS B J, DOBIN A, LI B, et al. Accuracy assessment of fusion transcript detection via read-mapping and de novo fusion transcript assembly-based methods[J]. Genome Biol, 2019, 20: 213.
[2] HAAS B, DOBIN A, STRANSKY N, et al. STAR-Fusion: Fast and Accurate Fusion Transcript Detection from RNA-Seq[EB/OL]. bioRxiv, 2017.
[3] KUMAR-SINHA C, TOMLINS S A, CHINNAIYAN A M. The emergence of gene fusions as biomarkers and therapeutic targets[J]. Cancer Discov, 2015, 5(1): 36-47.
[4] Detection of novel PPP1R1B::STARD3 fusion transcript in acute myeloid leukemia: a case report[J]. Journal of Medical Case Reports, 2023.
